原位杂交技术
目录
简介
原位杂交技术 (ISH) 是一种杂交,它使用标记的互补 DNA、RNA 或修饰的核酸链(即探针)将特定的 DNA 或 RNA 序列定位在组织的一部分或部分(原位)或 如果组织足够小(例如,植物种子、果蝇胚胎),则在整个组织(整装 ISH)、细胞和循环肿瘤细胞 (CTC) 中。 这与免疫组织化学不同,免疫组织化学通常将蛋白质定位在组织切片中。
原位杂交技术用于揭示特定核酸序列在染色体或组织中的位置,这是了解基因的组织、调控和功能的关键步骤。 目前使用的关键技术包括用寡核苷酸和 RNA 探针(放射性标记和半抗原标记)与 mRNA 原位杂交、光学显微镜和电子显微镜分析、整体原位杂交、RNA 和 RNA 加蛋白质的双重检测, 和荧光原位杂交检测染色体序列。 DNA ISH 可用于确定染色体的结构。 例如,荧光 DNA ISH (FISH) 可用于医学诊断以评估染色体完整性。 RNA ISH(RNA 原位杂交)用于测量和定位组织切片、细胞、整体和循环肿瘤细胞 (CTC) 中的 RNA(mRNA、lncRNA 和 miRNA)。 原位杂交技术是由美国生物学家Mary-Lou Pardue和Joseph G. Gall发明的。
原位杂交的挑战
原位杂交技术是一种强大的技术,可用于识别组织切片中单个细胞内的特定 mRNA 种类,从而深入了解生理过程和疾病发病机制。 然而,原位杂交需要采取许多步骤,对每个检查的组织和使用的每个探针进行精确优化。 为了将目标 mRNA 保存在组织内,通常需要使用交联固定剂(如甲醛)。
此外,组织切片的原位杂交要求组织切片非常薄,通常厚度为 3 µm 至 7 µm。 为原位杂交处理准备组织切片的常用方法包括用低温恒温器或 Compresstome 组织切片机切割标本。 低温恒温器将新鲜或固定的组织浸入液氮中进行速冻。 然后将组织包埋在称为 OCT 的冷冻介质中,并切下薄片。 障碍包括在组织上产生可能干扰正确 mRNA 染色的冻结伪影。 Compresstome 无需冷冻过程即可将组织切成薄片; 自由漂浮的部分在包埋在琼脂糖中以确保稳定性后被切割。 这种方法避免了冷冻组织,从而避免了相关的冷冻伪影。 一旦完成,该过程是xxx的且不可逆的。
过程
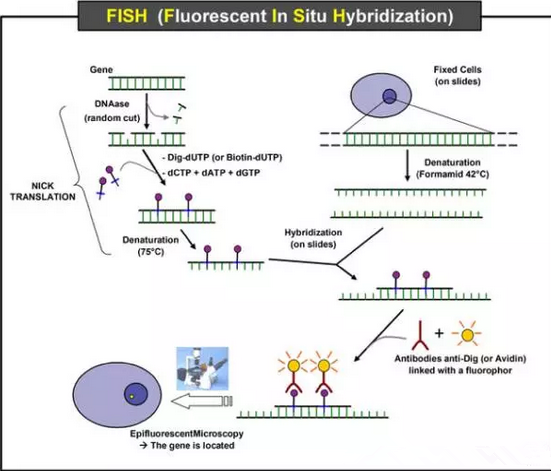
对于杂交组织化学,样本细胞和组织通常经过处理以将目标转录物固定到位并增加探针的可及性。 如上所述,探针要么是标记的互补 DNA,要么是现在最常见的互补 RNA(核糖核酸探针)。 探针在高温下与目标序列杂交,然后洗掉多余的探针(在未杂交的情况下,在使用 RNase 预先水解后,过量的 RNA 探针)。 可以操纵温度、盐和/或洗涤剂浓度等溶液参数,以消除任何不相同的相互作用(即,只有精确的序列匹配将保持绑定)。 然后,分别使用放射自显影术、荧光显微镜或免疫组织化学对用放射性、荧光或抗原标记的碱基(例如地高辛)标记的探针在组织中进行定位和定量。 ISH 还可以使用两个或多个标记有放射性或其他非放射性标记的探针来同时检测两个或多个转录本。
另一种技术,分支 DNA 测定,可用于 RNA(mRNA、lncRNA 和 miRNA)原位杂交测定,具有单分子敏感性,无需使用放射性。 这种方法(例如 ViewRNA 分析)可用于在一次分析中可视化多达四个靶标,并且它使用获得专利的探针设计和 bDNA 信号放大来生成敏感和特定的信号。 样本(细胞、组织和 CTC)被固定,然后进行处理以允许 RNA 靶标可及性(RNA 去掩蔽)。 目标特异性探针与每个目标 RNA 杂交。 随后的信号放大取决于相邻探针(在 RNA 靶标上并排结合的单个寡核苷酸 [oligos])的特异性杂交。 一个典型的目标特异性探针将包含 40 个寡核苷酸,产生 20 个寡核苷酸对并排结合在目标上以检测 mRNA 和 lncRNA,以及 2 个寡核苷酸或一对用于 miRNA 检测。 信号放大是通过一系列顺序杂交步骤实现的。 前置放大器分子与目标特异性 RNA 上的每个寡核苷酸对杂交。